1. 階層ベイズモデル
階層ベイズモデルを線形回帰に適用してみる。
1.1 まずは普通に推定…
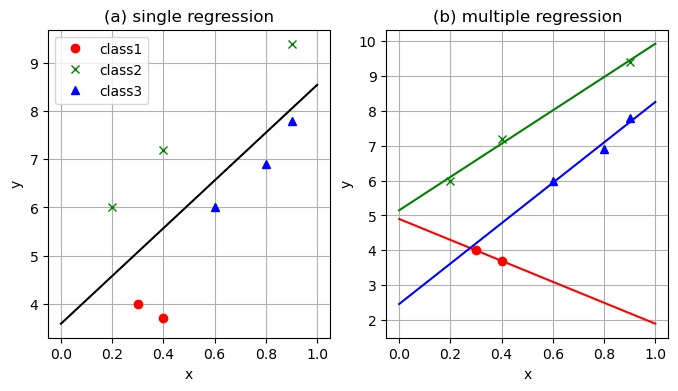
異なるクラスの観測値があるとき、これらを一括で標本とした場合と、クラスごとに標本とした場合の結果を比較してみる。
using Distributions, PyPlot, ForwardDiff, LinearAlgebra # n次元単位行列 eye(n) = Diagonal{Float64}(I, n) ###### # グラフの諸設定を行う関数 function set_options(ax, xlabel, ylabel, title; grid = true, gridy = false, legend = false) ax.set_xlabel(xlabel) ax.set_ylabel(ylabel) ax.set_title(title) if grid if gridy ax.grid(axis = "y") else ax.grid() end end legend && ax.legend() end ###### ######################## ### 階層ベイズモデル ### ######################## ### 全体傾向から個別傾向へと適合させていく ### 3クラスのデータに適用してみる # 学習データ X_obs = [] Y_obs = [] # 観測値 push!(X_obs, [0.3, 0.4]) push!(Y_obs, [4.0, 3.7]) push!(X_obs, [0.2, 0.4, 0.9]) push!(Y_obs, [6.0, 7.2, 9.4]) push!(X_obs, [0.6, 0.8, 0.9]) push!(Y_obs, [6.0, 6.9, 7.8]) # 観測値を可視化 fig, ax = subplots() ax.plot(X_obs[1], Y_obs[1], "or", label = "class1") ax.plot(X_obs[2], Y_obs[2], "xg", label = "class2") ax.plot(X_obs[3], Y_obs[3], "^b", label = "class3") set_options(ax, "x", "y", "data", legend = true) ###### # 線形回帰のパラメータ推定値 function linear_fit(Y,X) N = length(Y) w₁ = sum((Y .- mean(Y)) .* X) / sum((X .- mean(X)) .*X) w₂ = mean(Y) .- w₁*mean(X) w₁,w₂ end ###### ############################## ### 普通の回帰をやってみる ### ############################## # 回帰の実行 w₁,w₂ = linear_fit(vcat(Y_obs...), vcat(X_obs...)) # 個々の回帰 w₁s = [] w₂s = [] for i in 1:3 w₁_tmp, w₂_tmp = linear_fit(Y_obs[i], X_obs[i]) push!(w₁s, w₁_tmp) push!(w₂s, w₂_tmp) end # 可視化範囲 xs = range(0, 1, length = 1000) fig, axes = subplots(1,2,figsize = (8,4)) # 単一の回帰 axes[1].plot(xs, w₁ * xs .+ w₂, "-k") # 個別の回帰 axes[2].plot(xs, w₁s[1] .* xs .+ w₂s[1], "-r") axes[2].plot(xs, w₁s[2] .* xs .+ w₂s[2], "-g") axes[2].plot(xs, w₁s[3] .* xs .+ w₂s[3], "-b") # データの可視化 for ax in axes colours = ["or", "xg", "^b"] labels = ["class1", "class2", "class3"] for i in 1:3 ax.plot(X_obs[i], Y_obs[i], colours[i], label = labels[i]) end end set_options(axes[1], "x", "y", "(a) single regression", legend = true) set_options(axes[2], "x", "y", "(b) multiple regression", legend = false)
データのプロット

推定結果
1.2 階層ベイズモデルによる線形回帰
線形回帰を階層ベイズモデルとして書き直すことを考える。
をクラス
における標本数とし、同時分布でモデルを書き直すと
と書ける。ここではそれぞれ傾きおよび切片に対するハイパーパラメータであり、ここでは
と設定する。具体的にはとする。
ハイパーパラメータが生成されると、それに基づいて各クラスの傾きパラメータ
および切片パラメータ
がそれぞれ
に従って生成される。尤度の部分では、各クラスにおいて、生成されたパラメータを用いて平均値
が決まり、そこに誤差項が加わることで出力値
が生成される。
using Distributions, PyPlot, ForwardDiff, LinearAlgebra # n次元単位行列 eye(n) = Diagonal{Float64}(I, n) ###### # グラフの諸設定を行う関数 function set_options(ax, xlabel, ylabel, title; grid = true, gridy = false, legend = false) ax.set_xlabel(xlabel) ax.set_ylabel(ylabel) ax.set_title(title) if grid if gridy ax.grid(axis = "y") else ax.grid() end end legend && ax.legend() end ### 対数同時分布の設計 # メモリの都合から@viewsとする @views hyper_prior(w) = logpdf(Normal(0, 10.0), w[1]) + logpdf(Normal(0, 10.0), w[2]) @views prior(w) = sum(logpdf.(Normal.(w[1], 1.0), w[3:5])) + sum(logpdf.(Normal.(w[2], 1.0), w[6:8])) @views log_likelihood(Y, X, w) = sum([sum(logpdf.(Normal.(Y[i], 1.0), w[2+i].*X[i] .+ w[2+i+3])) for i in 1:3]) log_joint(w, X, Y) = hyper_prior(w) + prior(w) + log_likelihood(Y_obs, X_obs, w) params = (Y_obs, X_obs) ulp(w) = hyper_prior(w) + prior(w) + log_likelihood(w, params...) ### ### Hamiltonia Monte Carlo # Hamiltonian Monte Carlo method function HMC(log_p_tilde, μ₀; maxiter::Int64, L::Int64, ε::Float64) # leapfrog による値の更新 function leapflog(grad, p_in,μ_in, L, ε) μ=μ_in p = p_in + 0.5 *ε*grad(μ) for l in 1: L-1 μ +=ε*p p += ε*grad(μ) end μ += ε*p p += 0.5 *ε*grad(μ) p, μ end # 非正規化対数事後分布の勾配関数 grad(μ) = ForwardDiff.gradient(log_p_tilde, μ) # サンプルを格納する配列 D = length(μ₀) μ_samples = Array{typeof(μ₀[1]), 2}(undef, D, maxiter) # 初期サンプル μ_samples[:, 1] = μ₀ # 受容されたサンプル数 num_accepted = 1 for i in 2:maxiter # 運動量の生成 p_in = randn(size(μ₀)) # リープフロッグ p_out, μ_out = leapflog(grad, p_in, μ_samples[:, i-1], L, ε) # 比率rの対数を計算 μ_in = μ_samples[:, i-1] log_r = (log_p_tilde(μ_out) + logpdf(MvNormal(zeros(D),eye(D)), vec(p_out))) - (log_p_tilde(μ_in) + logpdf(MvNormal(zeros(D),eye(D)),vec(p_in))) # 確率rでサンプル受容 is_accepted = min(1, exp(log_r)) > rand() new_sample = is_accepted ? μ_out : μ_in # 新サンプルを格納 μ_samples[:,i] = new_sample # 受容された場合、合計を加算する num_accepted += is_accepted end μ_samples, num_accepted end ### function inference_wrapper_HMC(log_joint, params, w_init; maxiter::Int64 = 100_000, L::Int64 = 100, ε::Float64 = 1e-1) ulp(w) = log_joint(w, params...) HMC(ulp, w_init; maxiter = maxiter, L = L, ε = ε) end ### # HMCの初期設定 maxiter = 1000 w_init = randn(8) # 推定の実施 param_posterior, num_accepted = inference_wrapper_HMC(log_joint, params, w_init, maxiter = maxiter, L =100, ε=0.01) # 予測分布の可視化 fig, axes = subplots(1,3,sharey = true, figsize = (12,4)) for i in 1:3 for j in 1:size(param_posterior, 2) w₁,w₂ = param_posterior[[2+i,2+i+3],j] axes[i].plot(xs,w₁.*xs.+w₂, "c-", alpha = 0.1) end set_options(axes[i], "x", "y", "class $(i)") end colours = ["or","xg","^b"] for i in 1:3 axes[i].plot(X_obs[i],Y_obs[i],colours[i]) end tight_layout()

